|
Bonjour,
Plusieurs fils de discussion sur le forum du Naturaliste m'ont incité à écrire une série d'articles consacrés à la biologie moléculaire. Deux raisons au moins justifient, selon moi, leur présence sur le portail de notre site préféré : d'une part, cette discipline est au cœur de bien des débats de société actuels auxquels les naturalistes amateurs que nous sommes sont particulièrement sensibles (santé, environnement, agroalimentaire) et, d'autre part, son influence s'étend désormais jusque dans un domaine qui nous rassemble pour ainsi dire tous : le vivant, sa classification, son évolution et son utilisation par l'Homme.
Sachant que la formation scientifique des uns et des autres peut être plus ou moins étendue, et ne voulant perdre personne en route, j'ai décidé de découper mon propos en plusieurs courts articles et les proposer à la lecture sous forme de feuilleton. J'ai essayé de limiter le jargon à chaque fois que c'était possible sans nuire à la clarté de l'exposé. De même, le ton employé est généralement décontracté car il s'agit plus d'apporter des informations générales que d'écrire un cours de fac... Enfin, toutes les remarques ou suggestions visant à améliorer cet article (et les prochains à paraître) sont les bienvenues.
Bonne lecture !
Voici notre feuille de route :
- Épisode 1 - Généralités : ADN, génomes et gènes
- Épisode 2 - Techniques usuelles de biologie moléculaire
- Épisode 3 - Premières applications au laboratoire
- Épisode 4 - Agrobacterium tumefaciens et la transgenèse végétale
- Épisode 5 - La transplastomique, vers des OGM propres ?
L'expérience montre qu'il est difficile de comprendre le fonctionnement d'un mécanisme subtil en adoptant une approche statique. Bien au contraire, agir sur les rouages et perturber ainsi la mécanique interne aide à percer les mystères. C'est pourquoi, après avoir élucidé la structure de l'ADN, les biologistes moléculaires ont été amenés à développer leurs propres outils pour comprendre le fonctionnement des gènes. Quelques-uns de ces outils reposent sur des astuces assez géniales qu'il faut porter au crédit de leurs inventeurs, mais tous, sans exception, détournent un mécanisme naturel pour l'utiliser dans le contexte des expérimentations de laboratoire. Paradoxalement, ces outils ont eu pour effet de rendre la discipline plutôt abstraite car, finalement, on trouve toujours un intermédiaire entre le chercheur et son sujet d'étude. Pour casser cette impression, j'ai essayé, chaque fois que c'était possible, de faire des analogies avec des outils d'utilisation courante.
Attention, cette partie me paraît d'un abord plus difficile que la précédente. En particulier, si vous n'avez pas compris le principe de l'orientation des brins d'ADN, le texte ci-dessous risque de paraître cryptique. N'hésitez pas à retourner jeter un œil à l'épisode 1. Je sais aussi que je n'ai pas beaucoup développé certains points. Je modifierai le texte suite à vos remarques afin de le rendre plus clair et plus lisible.
La photocopieuse : réaction en chaîne par polymérase (PCR)
Une séquence d'ADN donnée (que ce soit un gène, un promoteur ou tout autre chose) est difficile à étudier telle quelle. Il est intéressant de la copier pour l'étudier hors de son contexte. C'est à Kary Mullis que revient le mérite d'avoir inventé la « photocopieuse d'ADN » ou PCR (polymerase chain reaction). Voici son principe : on souhaite recopier en masse une séquence donnée (en rouge sur le dessin ci-dessous). On va donc choisir 2 courtes séquences (de 18 à 25 bases en général) situées de part et d'autre de la séquence d'intérêt : ici nous avons choisi TATCAACCCCAAAACTTT et GAGACGAATAAGGTCC. Remarquez que ces deux séquences sont en orientation inverse : la première est choisie dans le sens 5'->3' et se fixe donc sur le brin complémentaire orienté de 3'->5', tandis que la seconde est choisie dans le sens 3'->5' et se fixe donc sur le brin orienté de 5'->3'.
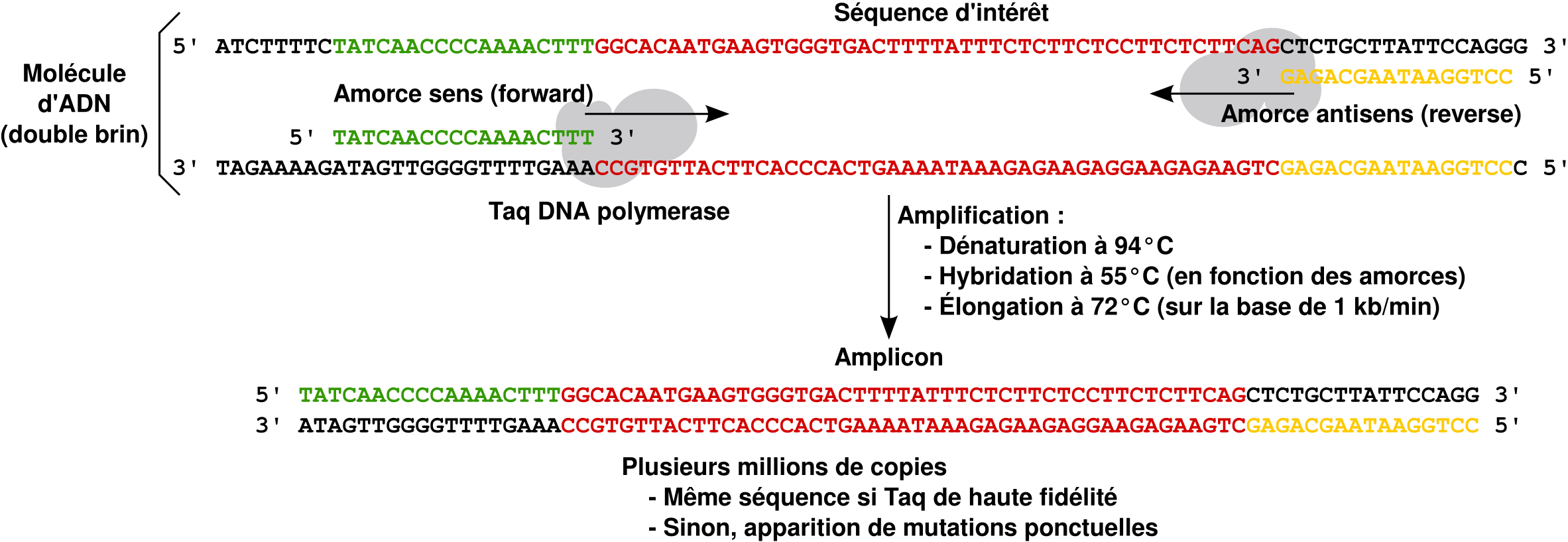
Pourquoi un tel choix ?
En fait, les enzymes qui recopient l'ADN, les ADN polymérases, travaillent toutes en sens 5'->3', jamais en sens 3'->5'. En choisissant nos courtes séquences tête-bêche, on recopie donc la séquence qui nous intéresse (en rouge). En effet :
- La courte séquence 5'->3' se lie à son brin complémentaire (le brin 3'->5' si vous avez suivi) et permet de recopier le brin orienté 5'->3'.
- La courte séquence 3'->5' se lie aussi à son brin complémentaire (le brin 5'->3') et recopie ainsi le brin orienté 3'->5'.
Ces « courtes séquences » sont appelées des amorces (en anglais primers) car elles amorcent la copie d'une séquence. Le brin utilisé comme support pour la copie porte quant à lui le nom de matrice. Et si on répète l'opération un grand nombre de fois, on recopie des milliers de fois la séquence qui nous intéresse, et seulement elle. Chouette !
Concrètement, il y a quand même un problème : pour que les amorces se fixent sur leur matrice, il faut que les deux brins de l'ADN soient dissociés... dans le cas contraire, ça ne fonctionne pas. Or, dissocier les deux brins nécessite une température élevée proche de 100°C. Or la plupart des polymérases sont détruites par la chaleur. Alors, comment faire ? Tout le mérite de Kary Mullis est d'avoir utilisé une polymérase issue d'une bactérie thermophile du parc de Yellowstone, Thermus aquaticus. Sa polymérase, appelée Taq, est résistante à de hautes températures. La copie (ou amplification) peut donc s'effectuer par cycles de la façon suivante :
- D'abord, dissociation des deux brins d'ADN à haute température (généralement 94°C) : c'est l'étape de dénaturation.
- Ensuite, fixation des amorces sur les brins isolés à une température choisie en fonction de la séquence des amorces : c'est l'étape d'hybridation (annealing en anglais). Une température trop basse permet aux amorces de se fixer sur des séquences proches mais pas exactement identiques, ce qui peut conduire à des copies parasites (non spécifiques).
- Enfin, copie de la séquence à 72°C (optimum de fonctionnement de la Taq) sur la base d'une minute pour 1000 paires de bases. Aujourd'hui, il existe dans les laboratoires différentes Taq plus ou moins rapides et plus ou moins fidèles vis-à-vis de la séquence d'origine. Les plus rapides sont utilisées en routine pour vérifier la présence ou l'absence d'une séquence donnée, tandis que les plus fidèles servent lorsque l'on veut extraire une séquence pour l'utiliser dans un contexte différent (on veut alors éviter toute erreur de copie, car l'apparition de mutations peut changer le sens de la séquence !).
Vous imaginez bien qu'une Taq vraiment fidèle coûte cher... et que, quelle que soit sa fiabilité, il faut quand même envoyer les séquences recopiées à séquencer dans des entreprises (quelques noms en vrac : Cogenics, GATC...) pour s'assurer que tout s'est bien passé avant d'aller plus loin dans le travail.
Je voudrais terminer en précisant que les cycles successifs d'amplification sont réalisés par des automates appelés thermocycleurs. Une PCR classique dure entre 45 min et 1h30. Il s'agit clairement d'une technique utilisée en routine.
 Thermocycleur Thermocycleur
Le Meccano : les plasmides
Un plasmide est une molécule d'ADN, généralement circulaire, qui mène une vie autonome dans l'organisme qui l'accueille (presque toujours une bactérie, mais aussi quelquefois des levures). Les plasmides sont un élément important des transferts horizontaux de gènes entre populations bactériennes. Ce sont aussi de précieux outils pour les chercheurs qui les ont détournés à leur profit. En effet :
- Si l'on conserve l'origine de réplication qui confère au plasmide son autonomie vis-à-vis du microorganisme,
- mais que l'on enlève les gènes qu'il contient (ils peuvent être éventuellement responsables du pouvoir pathogène),
- on obtient une sorte de machine autonome dans laquelle on peut insérer les gènes qui l'on veut !
Pour savoir à quoi servent les plasmides, on a coutume de les représenter sous forme d'anneaux en annotant les éléments importants. On appelle cela la carte d'un plasmide. Il existe des logiciels dédiés à la manipulation de ces cartes, qui permettent de reproduire par informatique (on dit : in silico) les modifications effectuées au laboratoire. On crée ainsi de nouvelles cartes correspondant aux plasmides modifiés, et ainsi de suite... Je vous donne ci-dessous un exemple de carte de plasmide utilisé au laboratoire.

Pour qu'il soit aisé de savoir ce que contiennnent les plasmides, une nomenclature a été mise au point. Dans l'exemple ci-dessous, on peut voir la carte du plasmide pK7FWG2. Son nom se décompose comme suit : K pour résistance à la kanamycine, 7 pour terminateur 35S (souvenez-vous, un terminateur se trouve en fin de gène), F pour GFP (voir ci-dessous), WG pour Gateway "inversé" (une technologie développée par la société Invitrogen dont je n'en parle pas ici) et 2 pour promoteur 35S (promoteur fort et constitutif, je rappelle qu'un promoteur sert à réguler l'expression d'un gène). En clair, ce vecteur permet d'exprimer un gène en fusion avec la GFP en 3' du gène ("à la fin" de la protéine).
Les ciseaux : les enzymes de restriction
Les enzymes de restriction sont utilisées par les bactéries pour se défendre contre les attaques virales des phages. Elles reconnaissent des séquences d'ADN spécifiques et les coupent, tout simplement. Ces séquences ont la particularité d'être des palindromes. Par exemple, vous voyez ci-dessous qu'une enzyme de restriction appelée EcoRI (lire "Éco R un") reconnaît et coupe le palindrome 5'-GAATTC-3'. Le nom de l'enzyme dérive de la bactérie dont elle est issue. Par exemple, l'enzyme EcoRI dérivée d'Escherichia coli. Ces enzymes sont utilisées par les bactéries pour se protéger contre les virus qui les affectent, les phages.

Vous comprenez sans doute mieux pourquoi la carte du vecteur pK7FWG2 ci-dessus comporte de nombreuses annotations comme SmaI, SacI, HindIII ou encore XmaI et SacII. Ce sont tout simplement les emplacements des sites reconnus par ces enzymes ! La fréquence des sites dépend de leur longueur (souvent 6 paires de bases) mais aussi de la composition de l'ADN considéré.
Les mouchards : les gènes rapporteurs
Comment savoir à quel moment s'exprime un gène d'intérêt ? En général, le produit d'un gène est difficile à détecter... surtout quand on ne connaît pas sa fonction ! Pourtant, l'étude de son expression spatio-temporelle est susceptible d'apporter de précieuses informations... il faudrait disposer de « mouchards moléculaires ». Est-ce difficile ? À bien y réfléchir, pas vraiment, puisque nous avons déjà à notre disposition une photocopieuse, des ciseaux et de la colle. Il suffit de trouver des gènes dont le produit (protéine) est facile à détecter... comme les protéines fluorescentes ou certaines enzymes qui catalysent une réaction colorée ! Je vous en cite quelques-unes : la célèbre GFP et ses dérivés, la beta-glucuronidase, la beta-galactosidase, la luciférase, etc. La suite en images (cliquez sur les images pour accéder à la page dont elles sont issues) :
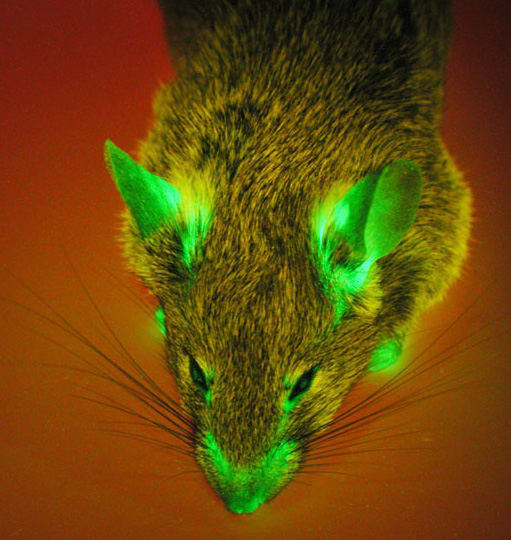 Souris transgénique GFP Souris transgénique GFP  Tamily Weissman, hippocampe de souris transgénique brainbow (x40) Tamily Weissman, hippocampe de souris transgénique brainbow (x40)
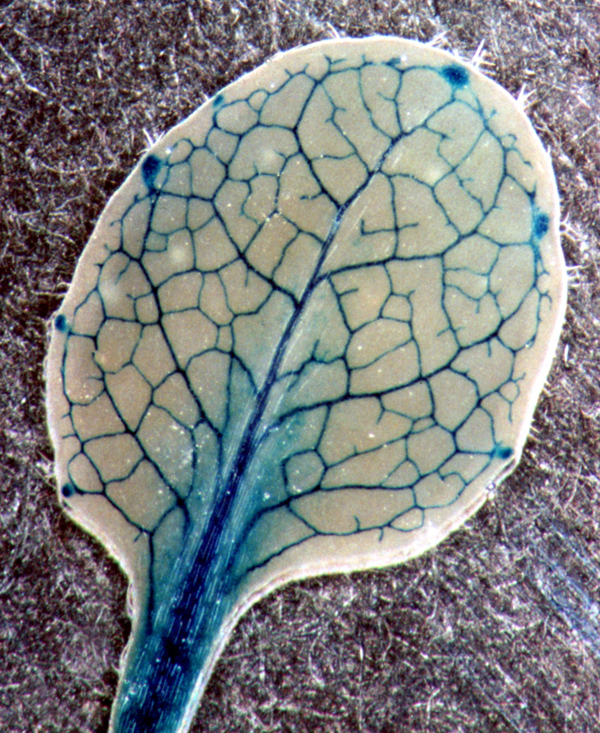 Feuille d'Arabidopsis thaliana transgénique exprimant le gène GUS sous le contrôle du promoteur RLK7. L'expression est localisée dans les tissus vasculaires. Feuille d'Arabidopsis thaliana transgénique exprimant le gène GUS sous le contrôle du promoteur RLK7. L'expression est localisée dans les tissus vasculaires.
Les gènes rapporteurs permettent de préciser l'expression spatio-temporelle des gènes, mais aussi de faire de la localisation subcellulaire en microscopie confocale. Par exemple, on peut déterminer si une protéine donnée se trouve dans le noyau, dans le réticulum endoplasmique ou dans d'autres compartiments.
Et si on mélangeait tout ?
Les quelques techniques que nous avons présentées peuvent être combinées pour obtenir des résultats vraiment intéressants. Par exemple, la réaction de PCR peut être effectuée directement sur des tissus après traitement approprié. Ce faisant, il est possible de mettre en évidence la présence d'un séquence dans un type cellulaire particulier. On parle de PCR in situ. Bien entendu, il existe bien d'autres techniques non évoquées ici. Je pourrais en parler davantage s'il y a des personnes intéressées. Il n'est pas toujours facile de fixer une limite judicieuse pour qu'un texte reste profitable au plus grand nombre. J'ai essayé de m'en tenir à l'essentiel.
Merci de votre lecture et à bientôt pour le troisième épisode !
Nous verrons à quoi servent concrètement ces techniques au laboratoire. 
Re: Génétique moléculaire - Épisode 2
--
Daniel
Thursday, 28 March 2019 18:39
Nous n'avons plus de message de Kevin depuis un moment.
Il doit être pris par ses études.
Essaie de le contacter par MP...
[quote="Daniel"]
Nous n'avons plus de message de Kevin depuis un moment.
Il doit être pris par ses études.
Essaie de le contacter par MP...
[/quote]
Re: Génétique moléculaire - Épisode 2
--
annkinn
Thursday, 28 March 2019 16:53
Bonjour,
Je découvre ces articles sur la biologie moléculaire fort intéressants, mais ne parvient pas à...
[quote="annkinn"]
Bonjour,
Je découvre ces articles sur la biologie moléculaire fort intéressants, mais ne parvient pas à trouver la suite comme annoncée dans le deuxième article.
Merci de votre réponse, bonne journée.
[/quote]
Re: Génétique moléculaire - Épisode 2
--
Maraussan
Friday, 17 May 2013 16:28
Formidable !
Seul bémol actuellement : il serait utile de valider ce qui est à l'origine des bugs de mise en forme en format PDF (via...
[quote="Maraussan"]
Formidable !
Seul bémol actuellement : il serait utile de valider ce qui est à l'origine des bugs de mise en forme en format PDF (via la fonctionnalité proposée en haut à droite du site Le Naturaliste).
[/quote]
Re: Génétique moléculaire - Épisode 2
--
nolwenn
Friday, 17 May 2013 10:17
Merci pour le partage !
[quote="nolwenn"]
Merci pour le partage !
[/quote]
Re: Génétique moléculaire - Épisode 2
--
Eddy
Saturday, 22 January 2011 18:15
Bonsoir le forum,
Voilà, l'italique a disparu. 
Désolé pour le...
[quote="Eddy"]
Bonsoir le forum,
Voilà, l'italique a disparu. 
Désolé pour le désagrément.
Cordialement,
Eddy
[/quote]
|